优雅的代价 - 记一次多分类分割的实现
经过上周的努力,嗅球识别模型的命中率已经比较可观了,但是误判率还是比较高。上周五老师给了我一份新数据,标注的是包括嗅球的周围白色区域的标签,类似下图蓝色部分:
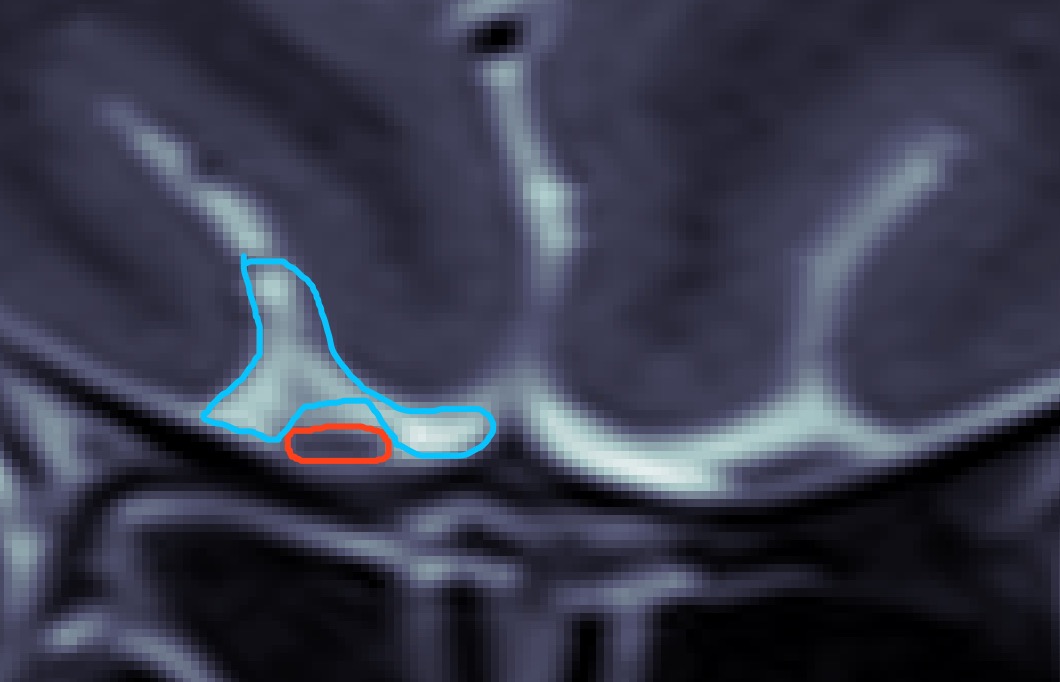
然后我产生了初步的利用白色区域标签的想法,即像训练嗅球那样训练出分割白色区域的模型,然后对于一张验证图片,分割出如上图所示的蓝色标签(白色区域)和红色标签(目标嗅球区域)。先前的嗅球区域会有一些错误并且偏离较大的预测,暂时简单的想法在识别出的白色区域下方设置一个阈值,超过这个阈值的预测认为是无效预测
接下来的问题就是像训练嗅球数据集一样训练白色区域的数据集了。我一开始的想法就是像训练嗅球一样,把nii数据转化成png之后一样的代码再训练一遍就是,但是仔细想想,既要训练嗅球,又要训练白色区域,这很明显是一个多分类问题,U-Net网络本来就支持多分类识别,那我是否可以在原来的代码基础上改动下输入的channel和class数呢?
于是尝试把嗅球和白色区域的原始nii数据叠加到RGB图片的红绿两个通道上去:
epi_img = nib.load(nii_path)
white_epi_img = nib.load(white_nii_path)
epi_img_data = []
epi_img_data.append(epi_img.get_fdata()) # 嗅球label
epi_img_data.append(white_epi_img.get_fdata()) # 白色区域label
_,_,frames = epi_img_data[0].shape #eg:a:896,b:896,frames:19(代表病人一有19张图)
for frame in range(frames):
label2write = np.zeros((896, 896, 3), np.uint8)
label_path = os.path.join(out_folder, names[frame] + ".png")
for i in range(2):
label = epi_img_data[i][:,:,frame]
label = label*255
label = np.rot90(label, k=1, axes=(1,0)) #图像旋转
label = np.fliplr(label) #图像翻转
if not label.shape[0] == 896:
label=cv2.resize(label,(896,896))
label2write[:,:,i+1] = label # 默认BGR通道,嗅球存G通道,白色区域存R通道
cv_imwrite(label_path, label2write) #保存整个完整嗅球相关组织人为标记标签的图片效果如下:

视觉上效果还是可以的,然后尝试把 U-Net 的 n_classes 参数换成2,跑起来提示了 torch 库里的很多有关 shape 的警告,大致是说可能无法正常广播。这个 warning 可不能忽略,广播机制出问题了的话最后模型可能会很奇怪,而且我也不觉得改了一个 n_classes 参数就行了。。。然后就是各种找原因,又是改 loss function 又是改最后一层的 sigmod 函数。就这样折腾了半天,仍然感觉有问题。最有帮助的还是 github 上的 这个issue。详细的看了一遍后发现,其中重点是数据集处理的不同,正确的做法应该是把原先二分类的 binary result mask 替换为标签为值的 gray result mask,比如说我要识别嗅球和白色组织,那我把嗅球编号为0,白色组织编号为1,标签中若某个像素为0则代表它是嗅球,为1则代表它是白色组织。而我是直接按照正常视觉效果的第一反应,多分类肯定是占用多个 channel 嘛,一种东西一个颜色,很符合直观感受。事实上那些多分类的模型效果图也是这样的,相信你一定见过类似这样的图:

说实话这张图技术层面上有点误导,但是视觉上确实能让人一眼看出分类的结果。不过反过来想想,也能找出几个理由说明RGB通道分类的不合理性,第一就是这样的话其实和之前的二分类一张 binary mask 没什么区别,只是把所有 class 的 mask 叠加到一块而已,没什么本质性的区别。第二就是我们被视觉迷惑了,虽然地图上有很多花花绿绿的色块能区分相邻区域,但是实际上 RGB 只能容纳 3 个分类而已,因为我们视觉上看到的黄色,是红色和绿色的叠加(例如上图中嗅球和白色组织重合的部分),到了计算机这里它就会认为这个像素既是嗅球,也是白色组织,但这显然是不可能的。如果我有 10 个分类,三通道的 RGB 根本无法满足需求,多通道的话则和理由一一样,并不能从根本上解决问题。
顺着这个思路想下去,本想跟着一步步改代码的,然而老师的代码和 github 上不太一样,只是网络结构一样,其他的例如对预测值的转化输出什么的,都要仔细研究明白了才好改。于是只好暂时放弃多分类代码的想法,先用不同的数据集训练白色区域的二分类。
冷静下来一想,或许单独训练白色区域的二分类才是比较好的选择,因为嗅球在 MRI 中的区域很小,基本就是两个小点,有效信息占比太小,所以之前训练都是把图像中间的320x320裁剪出来进行训练的;相比之下白色区域占比比较大。之前学姐的思路也是说,先大体确定嗅球所处的位置,然后再做精确化检测分割。上周我想的是取距离白色区域下方一定阈值内的嗅球,现在又有个新的想法:或许可以输入全图的白色组织数据训练找出白色组织的具体位置,然后根据白色组织的具体位置进行裁剪,再去跑之前320x320裁剪的嗅球模型,这样结合起来可能效果比较好,一定程度上解决了嗅球有效信息占比过小的问题,这样的话由于输入尺寸不同,反而分开跑两个二分类才是比较正确的选择了。
有的时候优雅的实现不一定更节省时间,也不一定表现最好。具体还是要多思考,根据需求的不同考虑怎样才能最优化,最优的才是最雅的
这样做的话生成的图像仍然是多通道的图像吧,一个类别一个通道。是不是有点麻烦?
请问如果Label是单通道灰度图,不同的灰阶表示类别的话,那么输出的类别数应该怎么写呢?是写通道数1还是类别数呢?
后来具体实现多分类的时候还是通过输出不同通道的形式,看起来确实有点麻烦,但是这样比较符合one-hot encoding的形式,即每个类别一个通道的话网络最后的输出就还是sigmoid之后的0~1范围,只是最后一层卷积的输出改为类别(通道)数。如果不同灰阶表示类别的话,最后的输出貌似就比较难处理了。可以试试直接把最后sigmoid那层删去,来回归具体的类别数值(灰阶)。这样做的话可能需要更改loss函数,参数可能也比较难调,但是感觉应该是可行的。通常这么做的似乎并不多,还是一个类别一个通道的输出比较常见